ここでは、当研究グループで開発した主にRhodococcus属細菌を宿主とした実験に使用している材料について紹介します。
誘導型発現ベクター(pTipベクター)
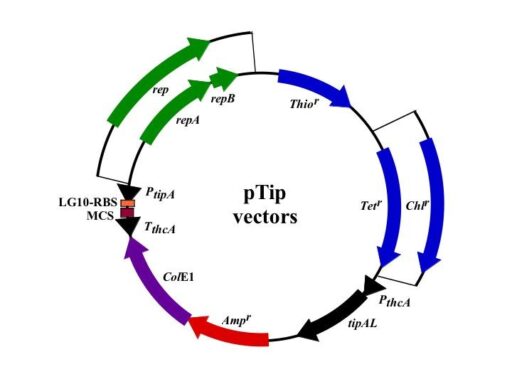
この誘導型発現ベクターは、抗生剤チオストレブトンによって発現が誘導されるtipAプロモーターを配したベクターで、10ng/mlという低濃度から発現誘導が可能です。また、発現は誘導後16時間程度で最大発現量を示します。用いる宿主によっては、4度での発現も可能です。ベクターには、2種類の複製開始起点を用意してあり、また、抗生剤耐性遺伝子も2種類用意してあります。プロモーター下流にはHis-tag融合タンパク質発現を容易にするための2種類のマルチクローニングサイトが用意されています。これらの組合せにより計8種類のベクターが構築されています。
参考文献
- Nakashima, N. & Tamura, T. (2004) Biotechnol. Bioeng., 86(2), 136-148.
- Nakashima, N. & Tamura, T. (2004) Appl. Environ. Microbiol., 70(9), 5557-5568.
構成型発現ベクター(pNitベクター)
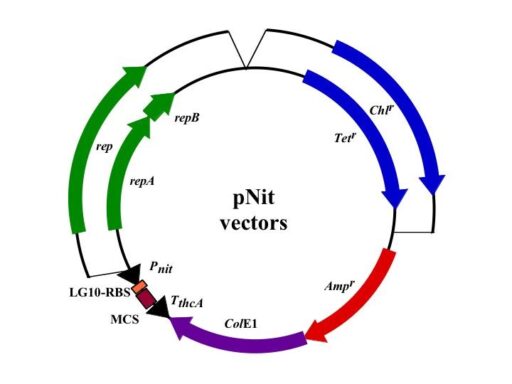
tipAプロモーターを改良してつくられた構成型プロモーター、nitプロモーターは、誘導剤を必要としない発現系に利用できます。発現量は、tipAプロモーターを使用した場合のほぼ半分程度です。ベクターの種類は、pTipベクターと同様計8種類用意されています。
参考文献
- Nakashima, N. & Tamura, T. (2004) Biotechnol. Bioeng., 86(2), 136-148.
- Nakashima, N. & Tamura, T. (2004) Appl. Environ. Microbiol., 70(9), 5557-5568.
トランスポゾンベクター
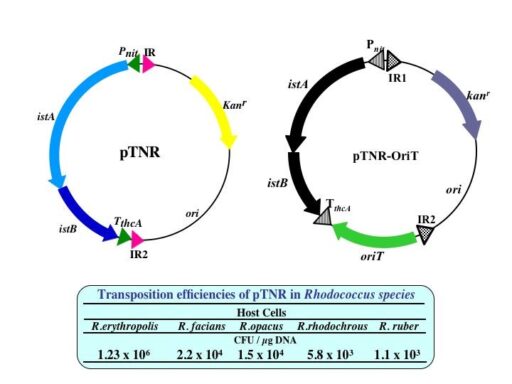
R. erythropolis由来挿入配列IS1415を利用したトランスポゾンベクター(pTNR)です。本来istABを挟むようにコードしていた反復配列(IR)を逆向きにかつ抗生剤耐性遺伝子と大腸菌用クローニングベクターのoriを挟み込むように構築しました。従って、このベクターは、Rhodococccus属細菌内における自律複製が困難であり、IR1とIR2に挟まれた領域がゲノムに挿入されることになります。ゲノムへの挿入は、任意の5-7塩基配列(9割は6塩基配列)を認識して転移しますので、ランダム変異ライブラリー構築に利用する事が出来ます。また、接合による転移を可能にしたoriTを導入したベクター(pTNR-OriT)も構築しています。その結果、ロドコッカス属放線菌以外の放線菌にも使用することが可能です。
また、DNA転移位置を容易に同定できるのもこのベクターの特徴です。本ベクターによりDNAが挿入されたゲノムを精製し任意の制限酵素で消化、閉環化した後、大腸菌に形質転換する事で転移部位周辺領域を含むプラスミドとして精製する事が可能です。
本ベクターは、ゲノムへ遺伝子を挿入できる事から、応用例として発現カセットをゲノムに挿入したタンパク質発現系も構築する事が可能です。導入する抗生剤耐性遺伝子の数を増やす事で、ゲノムに挿入するDNAの数も同様に増やす事が可能です。現在、4種の異なる抗生剤耐性遺伝子を導入したトランスポゾンベクターが構築されています。

更に、外来遺伝子を多コピー同時挿入可能なトランスポゾンベクターを開発しました。
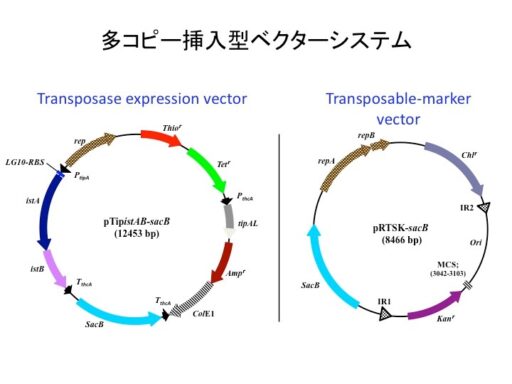
開発したシステムは、上図に示すベクター2種類を宿主細胞に同時に形質転換して使用するものです。左側は、転移に必要なトランスポゼースとそのヘルパータンパク質を発現するベクターで、発現誘導剤の添加により発現が誘導されます。一方、右側のベクターは転移マーカーを有するベクターで、IR1とIR2に挟まれた領域にカナマイシン耐性遺伝子とマルチクローニングサイトと大腸菌用のクローニングベクターのoriをコードしています。両ベクターは、上述したベクターと違って細胞内に安定に保存されるようにロドコッカス属細菌用ベクターの複製開始起点を持ちます。
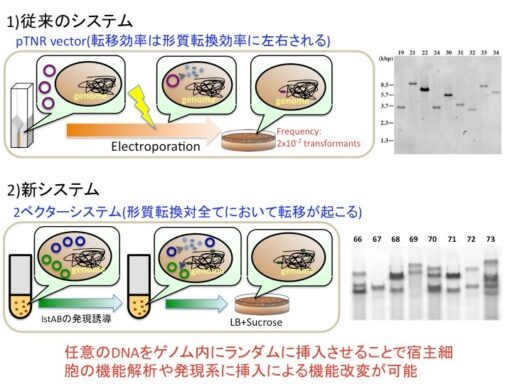
この両ベクターを用いたシステムを従来のシステムと比較したのが上図です。従来のシステムは、ベクターをエレクトロポーレーションなどで形質転換した後に、ベクターからトランスポゼースが発現して転移が引き起こされます。pTNRは、従来の系に比べると形質転換体50個に対して1個の割合で変異体を取得できる効率の良い転移効率を示しますが、変異体の取得数は、形質転換体の絶対数に依存するため形質転換効率などによって大きく作用されます。また、挿入されるDNAは1箇所のみです(上図右)。一方、新システムは、両ベクター共に細胞内に複数コピー安定に保持されます。そして、誘導剤を添加しない限り転移が起こらないので、両ベクターの形質転換体をいくらでも増殖させることが出来ます。そして、一旦トランスポゼースやヘルパータンパク質が発現すると、IRに挟まれた領域の転移が起こります。そして、細胞内にはベクターがマルチコピー存在するので、一旦転移が終了しても別の転移ベクターから転移が誘導されます。結果的に、培養した細胞全てに対して1から複数コピーの外来遺伝子をランダムに挿入することが可能になります(下図右)。このことより、容易に大量の変異体を取得できることのみならず、1遺伝子や1オペロンの変異や欠失だけでは見えなかった表現型に対する機能改変が可能になる可能性があります。
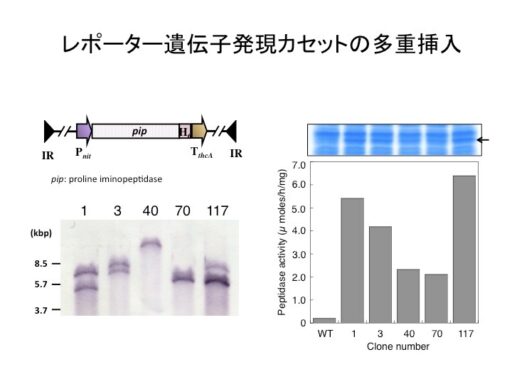
更に、この新システムを発現カセットの多コピー挿入に応用したのが上図です。レポーター遺伝子であるPIP(プロリンイミノペプチダーゼ)を構成型プロモーター支配下に連結した発現カセットを転移マーカーを有するベクターに組み込み宿主細胞ゲノムに対し多重挿入を試みました。その結果、発現カセットは1から3コピーまで挿入され(左下図)、そのコピー数に応じてPIPの発現量(右上図)ならびに活性が上昇(右下図)することが判明しました。このことは、上記システムを使えば、細胞内で発現ベクターを保持せずかつ発現量を制御可能なタンパク発現システムを構築出来ることが判明しました。この技術は、タンパク質の発現のみならず代謝工学に応用することが可能であると考えられます。
参考文献
- Sallam, K. I., Mitani, Y. & Tamura, T. (2006) J. Biotech., 121(1), 13-22.
- Sallam, K. I., Tamura, N. & Tamura, T. (2007) Gene, 386(1-2), 173-182.
- Sallam, K. I., Tamura, N., Imoto, N. & Tamura, T. (2010) Appl. Env. Microbiol., 76(8), 2531-2539.
リゾチーム感受性変異株
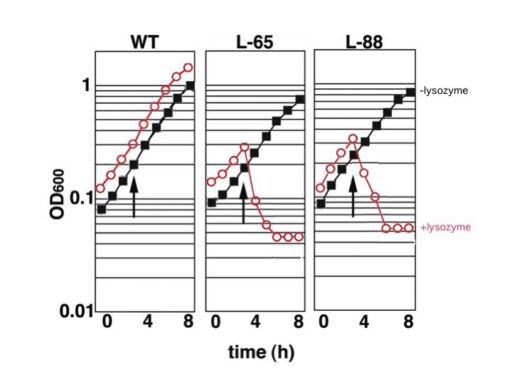
UV照射により点突然変異を導入したライブラリーから、リゾチームに対する感受性の高いR. eryhthropolis L-88とL-65株を単離しました。通常、野生株は1mg/ml以上のリゾチームに耐性を示しますが、単離した変異株は、12.5µg/mlで溶菌が起こる感受性株です。この菌株を利用する事で、細胞破砕が容易になりタンパク質の抽出・精製効率が著しく改善されます。なお、本菌における形質転換効率やタンパク質発現効率には野生株と何ら遜色ない事を確認しています。
参考文献
- Mitani, Y. et al., (2005) J. Bacteriol., 187(8), 2582-2591.
ロドコッカス細胞に孔を形成させた高効率物質変換系の開発
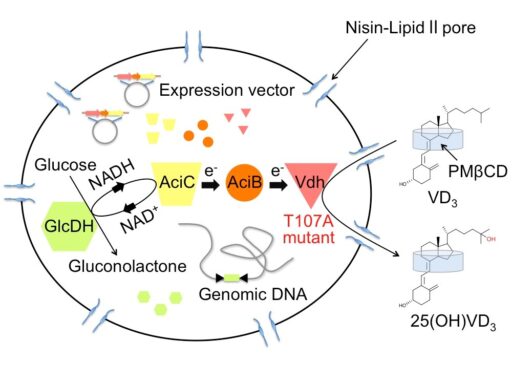
微生物細胞を利用した物質変換系において、基質の膜透過効率は反応効率に大きな影響を与えます。そこで、私たちは抗菌物質ナイシンを用いて細胞膜に穴(孔)を開けることにより、物質の細胞膜透過効率を飛躍的に高めた細胞変換系を開発しました。ナイシンはLactococcus lactis由来の34アミノ酸からなる抗菌ペプチドで、食品添加物としても認可されている物質です。ナイシンの作用機序はよく研究されており、主にグラム陽性細菌の細胞膜に直径2-2.5nm程度の孔(pore)を形成し、最終的には溶菌させることで抗菌活性を示します。しかしながらロドコッカス属細菌は他のグラム陽性細菌よりも溶菌しにくい性質があり、添加するナイシン濃度を最適化することで、孔は形成されながらも細胞構造は維持したまま溶菌しないユニークな状態を作り出すことができます。ほとんどの酵素はこの孔を通り抜けることができない一方で、分子量の小さな基質分子は効率良く細胞膜を透過できるようになります。私たちはこのナイシン処理を行ったR. erythropolis死細胞を用いることで、高効率に水酸化ビタミンDを生産することに成功しました。また、この細胞は内部に保持される酵素が失活しない限り、遠心分離によって回収した上で、繰り返し物質変換反応に利用できることを確認しています。
参考文献
- Imoto N. et al., (2011) Biochem. Biophys. Res. Commun., 405(3), 393-398.
- Yasutake, Y. et al., (2013) ChemBioChem, 14(17), 2284-2291.
Rhodococcus erythropolisを用いた形質転換の標準的なプロトコル
Competent cellの作製
- シングルコロニーをLB培地に植菌し、28℃にて2〜3日震盪培養
- 培養液をよく懸濁し、100µLを10mLの新たなLB培地に植菌(バッフル付きフラスコが望ましい)
- OD600が1.0程度になるまで28℃にて震盪培養
- 遠沈管に培養液を回収し氷上にて30分静置
- 4℃、3000xgにて10分間遠心後、上清を捨て菌体ペレットを回収
- 菌体ペレットに10mLの純水(4℃)を加え、ピペッティングにより再懸濁
- 5、6の操作をさらに2回繰り返す(計3回)
- 菌体ペレットに10mLの10%glycerol(4℃)を加え、ピペッティングにより再懸濁
- 5と同様に遠心して菌体ペレットを回収
- 4mLの10%glycerol(4℃)を加え、ピペッティングにより再懸濁
- マイクロチューブに400µLずつ分注し、-80℃にて凍結・保管
Electroporation法によるプラスミドの導入
- competent cellを氷上にて融解
- 10~100ngのプラスミドDNA*を加えて、氷上30分静置
*プラスミドを大腸菌で増幅する際にはDH5a、XL1-blueなどを用いる - 2.5kV、25µF、400Ωにてパルス印加(2mmキュベットの場合)(1mmキュベットの場合は1.6kV、25µF、400Ω)
- 1mLのLBにパルス印加済み菌液を添加し、28℃にて1時間震盪培養
- 200µLを適当な抗生剤の入ったLB寒天培地に塗布
抗生剤の終濃度:tetracycline = 10µg/mL, chloramphenicol = 20µg/mL
(大腸菌でのプラスミド保持にはampicillin = 50µg/mL) - 28℃にてコロニーが出るまで静置培養(2~3日)
組換えタンパク質発現(pTipの場合)
- 発現ベクター導入シングルコロニーを適当な抗生剤を含むLB培地に植菌し、28℃にて定常期に入るころまで震盪培養(通常2~3日)
- 培養液をよく懸濁し、1mLを9mLの新たなLB培地に植菌(バッフル付きフラスコが望ましい)
- OD600が0.6~0.8になるまで28℃にて震盪培養
- 終濃度0.1~0.2µg/mLとなるようthiostrepton*を添加し、さらに16時間振盪培養を続ける*thiostreptonのstock solution は1mg/mL(in DMSO)とし-20℃保存
組換えタンパク質発現(pNit, pCpiの場合)
- pTipの場合と同様に1、2の操作を行い、そのまま24~48時間培養を続ける
細胞破砕液回収(リゾチーム感受性R. erythropolis L88株を宿主とした場合)
- 上記の手順で得られた10mLの培養液を4℃、3000xgにて10分間遠心後、上清を捨て菌体ペレットを回収*
*回収後、凍結融解操作を行えばこの後の溶菌効率はよくなる - ペレット融解後、適当なバッファー(通常はpH8.0付近のリン酸バッファー)1mLに懸濁
- 最濃度1mg/mL以上(濃度が高いほど溶菌効率はよくなる)となるようにリゾチームを添加し氷上で1時間放置
- 超音波法やガラスビーズ法により細胞破砕を行う(時間・出力等は機種に依存)
*試料温度が高くならないように注意する - 4℃、20,000xgにて遠心し上清を細胞破砕液として回収する
実験ツールとしての宿主やベクターリスト
Strain
Rhodococcus erythropolis L88
(lysozyme sensitive mutant of R. erythropolis JCM3201)
Expression vectors
| Name | promoter | Selection | Replication | MCS |
|---|---|---|---|---|
| pTipQT1 | tipA(inducible) | tetracycline | theta type | type1 |
| pTipQT2 | tipA(inducible) | tetracycline | theta type | type2 |
| pTipQT1 | tipA(inducible) | tetracycline | rolling circle | type1 |
| pTipQT2 | tipA(inducible) | tetracycline | rolling circle | type2 |
| pTipQT1 | tipA(inducible) | chloramphenicol | theta type | type1 |
| pTipQT2 | tipA(inducible) | chloramphenicol | theta type | type2 |
| pTipQT1 | tipA(inducible) | chloramphenicol | rolling circle | type1 |
| pTipQT2 | tipA(inducible) | chloramphenicol | rolling circle | type2 |
| Name | promoter | Selection | Replication | MCS |
|---|---|---|---|---|
| pNitQT1 | nit(constitutive) | tetracycline | theta type | type1 |
| pNitQT2 | nit(constitutive) | tetracycline | theta type | type2 |
| pNitQT1 | nit(constitutive) | tetracycline | rolling circle | type1 |
| pNitQT2 | nit(constitutive) | tetracycline | rolling circle | type2 |
| pNitQT1 | nit(constitutive) | chloramphenicol | theta type | type1 |
| pNitQT2 | nit(constitutive) | chloramphenicol | theta type | type2 |
| pNitQT1 | nit(constitutive) | chloramphenicol | rolling circle | type1 |
| pNitQT2 | nit(constitutive) | chloramphenicol | rolling circle | type2 |
| Name | promoter | Selection | Replication | MCS |
|---|---|---|---|---|
| pCpiQT1 | cpi(constitutive) | tetracycline | theta type | type1 |
| pCpiQT2 | cpi(constitutive) | tetracycline | theta type | type2 |
| pCpiQT1 | cpi(constitutive) | tetracycline | rolling circle | type1 |
| pCpiQT2 | cpi(constitutive) | tetracycline | rolling circle | type2 |
| pCpiQT1 | cpi(constitutive) | chloramphenicol | theta type | type1 |
| pCpiQT2 | cpi(constitutive) | chloramphenicol | theta type | type2 |
| pCpiQT1 | cpi(constitutive) | chloramphenicol | rolling circle | type1 |
| pCpiQT2 | cpi(constitutive) | chloramphenicol | rolling circle | type2 |
Transposon vectors
| Name | Selection | |
|---|---|---|
| pTNR | kanamycine | single copy |
| pTNR-KA | kanamycine | single copy |
| pTNR-CA | chloramphenicol | single copy |
| pTNR-AA | apramycine | single copy |
| pTNR-istAB-sacB | kanamycine | multiple copies |
| pRTSK-sabB |

