HIV-1 reverse transcriptase with Q151M/F115Y/Y116F/G112S/D113A/F160L/I159L (7MC)
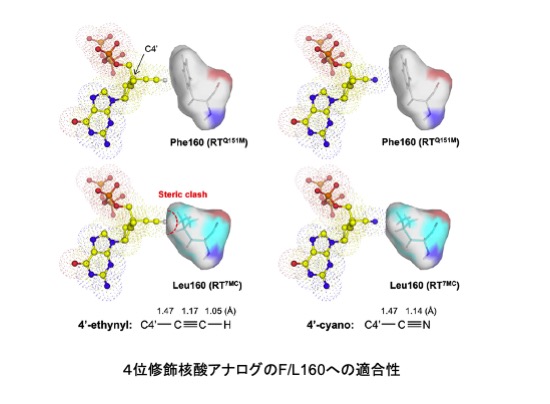
HIV RTの活性部位(薬剤結合部位)を形成するアミノ酸をHBV RTのアミノ酸にことごとく置換した多重変異体を作成したところ、残念ながらその多くはHIVの複製を阻害し、抗ウイルスアッセイによる薬剤感受性の評価ができませんでした。一方、組換え発現精製したそれら多重変異体は逆転写活性を保持していたので、その立体構造を解析し、変異により活性ポケットがどのように変化しているのかを観察しました。さらに、多重変異体7MCの立体構造に対して近年HIV/HBV治療薬として注目されている4位修飾核酸アナログをモデリングしたところ、ポケット底部を構築するPhe/Leu160の構造的差異により、4位シアノ/エチニルによる薬剤感受性の違いを合理的に説明することができた(図2)。RT 7MC構造は、種々の抗HBV核酸アナログのインシリコンモデリングを行うための新たな基準であり、新規抗HBV薬設計のために重要な構造情報を提供します。
関連論文
- Yasutake Y, Hattori SI, Tamura N, Matsuda K, Kohgo S, Maeda K & Mitsuya H. (2019) Active-site deformation in the structure of HIV-1 RT with HBV-associated septuple amino acid substitutions rationalizes the differential susceptibility of HIV-1 and HBV against 4ʹ-modified nucleoside RT inhibitors. Biochem. Biophys. Res. Commun., 509(4), 943-948. [PubMed] [PDB codes: 6ik9 & 6ika]

